Lloyd-Price, J., Abu-Ali, G. y Huttenhower, C. El microbioma humano sano. Genoma Med 8, 51 (2016). https://doi.org/10.1186/s13073-016-0307-y
Abstracto
Los seres humanos son prácticamente idénticos en su composición genética, sin embargo, las pequeñas diferencias en nuestro ADN dan lugar a una tremenda diversidad fenotípica en toda la población humana. Por el contrario, el metagenoma del microbioma humano (el contenido total de ADN de los microbios que habitan en nuestros cuerpos) es bastante más variable, ya que solo un tercio de sus genes constituyentes se encuentran en la mayoría de los individuos sanos. Por lo tanto, comprender esta variabilidad en el “microbioma saludable” ha sido un desafío importante en la investigación del microbioma, que se remonta al menos a la década de 1960, continuando a través del Proyecto Microbioma Humano y más allá. Catalogar los conjuntos necesarios y suficientes de características del microbioma que apoyan la salud, y los rangos normales de estas características en poblaciones sanas, es un primer paso esencial para identificar y corregir las configuraciones microbianas que están implicadas en la enfermedad. Con este objetivo, varios estudios a escala poblacional han documentado los rangos y la diversidad tanto de las composiciones taxonómicas como de los potenciales funcionales que normalmente se observan en los microbiomas de poblaciones sanas, junto con posibles factores impulsores como la geografía, la dieta y el estilo de vida. Aquí, revisamos varias definiciones de un “microbioma saludable” que han surgido, la comprensión actual de los rangos de diversidad microbiana saludable y brechas como la caracterización de la función molecular y el desarrollo de terapias ecológicas que se abordarán en el futuro.
Fondo
Los seres humanos han coevolucionado con los billones de microbios que habitan en nuestros cuerpos y que crean ecosistemas complejos, adaptativos específicos para cada hábitat del cuerpo, que están finamente sintonizados con la fisiología del huésped que cambia implacablemente. Las disbiosis en el microbioma se han asociado con numerosas enfermedades, incluida la enfermedad inflamatoria intestinal, la esclerosis múltiple, la diabetes (tipos 1 y 2), las alergias, el asma, el autismo y el cáncer [1-5]. Al igual que el concepto de patogenicidad de un solo taxón microbiano, la disbiosis de una comunidad microbiana puede ser difícil de definir, pero podría considerarse como una perturbación que se aparta de una ecología equilibrada [1] para prolongar, exacerbar o inducir un efecto perjudicial para la salud. Por lo tanto, encontrar características que distingan ampliamente los microbiomas sanos de los no saludables ayudará en el diagnóstico de enfermedades relacionadas con el microbioma y podría proporcionar nuevos medios para prevenir la aparición de enfermedades o mejorar el pronóstico. Se han propuesto muchas características potenciales comunes a los microbiomas sanos, incluidos los organismos prevalentes o las vías moleculares [6], así como las normas de ciertas propiedades ecológicas, como la diversidad o la estabilidad [7, 8]. Los microbiomas muestran regularmente un alto grado de diversidad interpersonal, incluso en ausencia de enfermedades [7, 9]. Esto complica la identificación de constituyentes microbianos simples o desequilibrios que causan enfermedad o reflejan un estado de enfermedad. Por lo tanto, la comprensión de las propiedades de un microbioma saludable y de las diferentes ecologías microbianas que se encuentran en ausencia de enfermedades manifiestas es, por lo tanto, un primer paso necesario para identificar y corregir las configuraciones microbianas que están implicadas en la enfermedad.
En esta revisión, usamos “sano” para referirnos a la ausencia de cualquier enfermedad manifiesta (como se define en [10], a menos que se especifique lo contrario para estudios particulares). La mayoría de los datos disponibles describen el microbioma intestinal y muchos de los hallazgos discutidos aquí son de esta área, aunque la mayoría de los principios se aplican a los hábitats microbianos en todo el cuerpo. Las primeras investigaciones sobre la ecología del microbioma buscaron identificar un conjunto “central” de taxones microbianos universalmente presentes en individuos sanos que carecen de fenotipos de enfermedad manifiesta, bajo la hipótesis de que la ausencia de tales microbios indicaría disbiosis [11]; Pero los estudios de la diversidad ecológica entre individuos sanos revelaron suficiente variación en la composición taxonómica del microbioma para hacer rápidamente que tal hipótesis sea poco probable [11, 12]. Incluso se encontró que los taxones compartidos, desde especies individuales hasta filos completos, variaban en abundancia en más de un orden de magnitud entre los individuos sanos [7, 11]. Por lo tanto, caracterizar un microbioma “sano” como un conjunto ideal de microbios específicos ya no es una definición práctica [2, 6].
Una hipótesis alternativa es la de un “núcleo funcional” saludable: un complemento de funciones metabólicas y otras funciones moleculares que son realizadas por el microbioma dentro de un hábitat particular, pero que no son necesariamente proporcionadas por los mismos organismos en diferentes personas [6]. Es posible que dicho núcleo deba estar presente como potencial genético (es decir, codificado dentro de los metagenomas del ADN) de la misma manera que el genoma humano no debe codificar mutaciones deletéreas graves para estar sano o puede necesitar ser expresado y bien regulado dentro de un individuo para que permanezca sano (es decir, debe estar codificado por metatranscriptomas de ARN o presente en forma de proteínas o productos de moléculas pequeñas). o, por supuesto, una combinación de los mismos. El núcleo funcional debe, por supuesto, incluir al menos las funciones de mantenimiento necesarias para la vida microbiana individual, que deben estar presentes genómicamente y expresarse correctamente; Curiosamente, estas propiedades también pueden incluir funciones específicas de los nichos de los microbios en el ecosistema humano. Tales funciones pueden incluir procesos que no son llevados a cabo por las células humanas y, por lo tanto, representan una base potencial para las relaciones simbióticas entre el huésped y la microbio. Un microbioma saludable puede caracterizarse además por su comportamiento a lo largo del tiempo [2, 8]; Intuitivamente, un microbioma asociado a la salud debe tener un grado de resiliencia a los cambios externos (por ejemplo, dietéticos o farmacéuticos) o internos (por ejemplo, relacionados con la edad o la deriva estocástica). Incluso si una estructura comunitaria en particular proporcionara todas las funciones básicas necesarias, sin esta resiliencia no podría garantizar estas funciones por mucho tiempo. Por lo tanto, la resistencia de un microbioma al estrés y la perturbación y su capacidad para recuperarse a un perfil funcional saludable después se encuentran entre las propiedades potenciales que caracterizan a un microbioma saludable [2, 13].
Aquí, revisamos la caracterización actual del microbioma saludable en términos de los residentes microbianos normales y sus funciones principales, propiedades ecológicas y dinámica temporal. Concluimos identificando las principales preguntas pendientes y las direcciones de investigación en este campo y especulamos sobre sus soluciones e impacto. Una combinación de los avances tecnológicos recientes y la actividad dentro del campo ha impulsado un aumento del interés en el microbioma humano en la salud y la enfermedad (Tabla 1) y, por lo tanto, esta revisión tiene como objetivo resumir la variedad de perspectivas actuales sobre lo que puede constituir un microbioma saludable.
Tabla 1 Diversidad de investigaciones recientes sobre el microbioma, que se han centrado principalmente en el intestino
| Publicaciones | ||
|---|---|---|
| Letra chica | Todo | 2011–2016 |
| Intestino | colon | intestinal | 17,546 | 10,707 |
| 0ral | boca | Lengua | diente | subgingival | supragingival | 4843 | 2089 |
| Urogenital | vaginal | Peneal | 1477 | 706 |
| Piel | cutáneo | 1372 | 754 |
| Vía aérea | pulmón | 764 | 524 |
| Placenta | leche materna | 702 | 426 |
| Ocular | ojo | 152 | 82 |
- Número de resultados obtenidos mediante la búsqueda de “(microbioma | Microbiota | microflora) (<Terms>)” en PubMed (consultado el 31 de marzo de 2016)
Nuestra comprensión evolutiva del microbioma saludable
Los primeros estudios trataron de identificar el conjunto normal de microbios que colonizan a las personas sanas, principalmente en el intestino, mediante el cultivo y la caracterización de las propiedades fisiológicas. Estos estudios destacan mejor los organismos que crecen bien en el entorno de laboratorio, como la Escherichia coli. Este sesgo llevó a la percepción de que la E. coli es un miembro abundante y prevalente del microbioma intestinal humano [14]. La introducción de técnicas estrictamente anaeróbicas en la década de 1970 permitió la recuperación de más de 300 especies bacterianas solo del intestino [15]; Además, el conteo de células viables dentro de diluciones seriadas estandarizadas en medios selectivos permitió la cuantificación de estas especies. Un resumen de cuatro grandes estudios de esta época [12] que analizaron muestras de heces de 141 estadounidenses con diferentes dietas encontró que las bacterias del género Bacteroides y los cocos anaeróbicos eran prevalentes y abundantes, mientras que el género Clostridium era ubicuo en menor abundancia, aunque no se observó una sola especie (como se definía entonces) en todos los sujetos. Otras bacterias prevalentes pero de menor abundancia incluían miembros de los géneros Bifidobacterium, Eubacterium, Lactobacillus y Streptococcus, así como anaerobios facultativos como Escherichia.
Ya se sospechaba en ese momento que un gran número de especies microbianas asociadas a los humanos seguían sin descubrirse, y un estudio estimó la presencia simultánea de unas 400 especies microbianas en un colon sano [16, 17]. Sin embargo, los exigentes requisitos de algunos microbios y la naturaleza intensiva en mano de obra del trabajo requerido para cultivarlos presentaron una barrera significativa para su descubrimiento [12]. Además, no todos los microbios pueden distinguirse bien como especies o cepas mediante el cultivo solo en medios selectivos; por ejemplo, las diferentes especies de Bacteroides de alta abundancia son particularmente difíciles de desentrañar [12, 17]. Además, estos estudios de la composición de la comunidad eran aún más difíciles de extender a los microbios no bacterianos, como los virus y los hongos, y eran aún menos prácticos para los estudios de hábitats corporales que son menos ricos en microbios que el intestino. Se requerían nuevos métodos para estudiar estos aspectos del microbioma saludable.
Las técnicas independientes del cultivo, como la secuenciación del ADN [18] y la hibridación fluorescente in situ (FISH) [19], están ahora muy extendidas y su democratización ha permitido interrogar directamente el contenido de ADN de las muestras microbianas [20]. Los primeros estudios que utilizaron FISH dirigido al gen de ARN ribosómico 16S sugirieron que al menos dos tercios de las bacterias intestinales en una cohorte de Europa occidental podrían atribuirse a un conjunto de seis grupos aproximadamente a nivel de especie/género: dos Bacteroides, dos Clostridium, Streptococcus/Lactococcus y Eubacterium rectale [19]. Desde entonces, esto ha demostrado ser optimista e, incluso en ese momento, se observó una gran variabilidad en las abundancias de estos grupos entre las muestras (desviaciones estándar de ~ 60-80 % de sus medias) [19].
Algunos de los primeros esfuerzos para secuenciar los genes de ARNr 16S directamente a partir de muestras mostraron que el 85-95 % de la abundancia bacteriana correspondiente a especies conocidas podría atribuirse a tres grupos bacterianos relacionados con Bacteroides, Clostridium cluster XIVa y Clostridium cluster IV [21, 22]. Los estudios de 16S también mostraron una gran diversidad en la composición taxonómica tanto entre personas sanas como entre sitios biogeográficos estrechamente vinculados dentro de una misma persona (como muestras de mucosas y heces [23]). Sin embargo, en todos estos estudios, la mayoría (75-80 %) de los grupos de secuencias no coincidían con ninguna especie documentada en ese momento [21-23], lo que explica gran parte de la subestimación de la diversidad en trabajos anteriores.
El advenimiento de la secuenciación masiva paralela (tecnologías de secuenciación de alto rendimiento) ha resuelto sustancialmente la composición taxonómica de esta “materia oscura” microbiana [24], aunque aún queda por caracterizar un porcentaje sorprendente de diversidad funcional (hasta el 50 % [25]), al igual que la composición de poblaciones no referenciales (que se analiza más adelante). Los hallazgos iniciales se hicieron eco de las grandes diferencias interpersonales, incluso entre gemelos [26], pero también implicaron la existencia de un conjunto de genes microbianos que son comunes a todos los individuos [26, 27]. Esto ayudó a sembrar el modelo de que, al igual que los genes de mantenimiento conservados en organismos individuales, un “microbioma central” puede definirse a nivel funcional en lugar de taxonómico [26, 27].
Cohortes basales a escala poblacional
Desde entonces, se han puesto en marcha proyectos a gran escala para caracterizar la diversidad de la composición microbiana y su potencial funcional, basándose en el rendimiento y la rentabilidad cada vez mayores de la secuenciación y otros ensayos moleculares. En 2010, el estudio Metagenomes del Tracto Intestinal Humano (MetaHIT) informó de metagenomas intestinales de muestras de heces de una cohorte de 124 adultos europeos (predominantemente «sanos»), que en ese momento superaban el volumen de secuenciación de todos los estudios anteriores sobre el microbioma en casi 200 veces [9]. En 2012, el Proyecto del Microbioma Humano (HMP, por sus siglas en inglés) informó de los resultados del perfil 16S en 242 adultos sanos de los Estados Unidos y la secuenciación metagenómica en un subconjunto de 139 individuos, con muestras que representaban 18 hábitats corporales distribuidos entre cinco áreas corporales principales [7]. Un gran estudio chino sobre la diabetes tipo 2 pronto contribuyó con 145 metagenomas intestinales adicionales, aproximadamente la mitad de los cuales procedían de controles no diabéticos [28]. Además, desde entonces, el consorcio MetaHIT ha seguido publicando nuevos metagenomas intestinales de adultos europeos [29-31]. En total, el número de microbiomas sanos a escala poblacional estudiados en el intestino y otras partes del cuerpo supera ahora los 2000 individuos que abarcan varios continentes.
Componentes típicos y diversidad del microbioma
Componentes bacterianos de un microbioma saludable
El ecosistema del colon ha sido el hábitat corporal más intensamente estudiado (Tabla 1), ya que cuenta con una notable diversidad entre las personas y una biomasa microbiana (recuento de células) que eclipsa a la de otros sitios corporales en más de un orden de magnitud [32]. En combinación con el aumento temprano de la secuenciación del gen 16S rRNA y las técnicas de cultivo anaeróbico, estas propiedades del intestino han llevado a un enfoque particularmente fuerte en la literatura sobre los residentes del microbioma intestinal bacteriano. Hasta la fecha se han caracterizado más de 1000 especies bacterianas intestinales [15], lo que proporciona una importante “lista de partes” de componentes bacterianos. Curiosamente, la filogenética molecular ha llevado a la reclasificación de muchas de estas especies en los últimos 20 años. De particular interés, las especies dentro de Bacteroides, anteriormente consideradas el género bacteriano más prevalente y abundante en el intestino, han sido reclasificadas en cinco géneros: Alistipes, Prevotella, Paraprevotella, Parabacteroides y Odoribacter, con trabajo molecular adicional basado en cultivos e independiente del cultivo en curso [15]. Se estima que entre 1000 y 1150 especies bacterianas prevalecían en los microbiomas intestinales de la cohorte MetaHIT, de las cuales cada persona portaba ~160 especies en promedio [9]. Los microbiomas intestinales sanos, evaluados mediante secuenciación, están dominados sistemáticamente por bacterias de dos filos, Bacteroidetes y Firmicutes [7, 9], aunque incluso cuando se considera este amplio nivel de clasificación, los individuos varían en más de un orden de magnitud en sus proporciones Firmicutes/Bacteroidetes [7]. Las bacterias prevalentes en las heces que se han identificado mediante técnicas moleculares han ampliado las listas anteriores para incluir bacterias de al menos ocho familias (Fig. 1a).
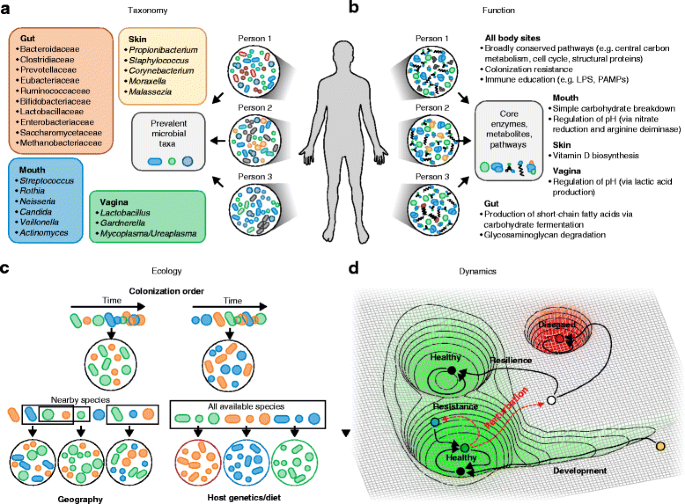
Posibles definiciones de un microbioma saludable: composición, función, dinámica y ecología. a Las primeras definiciones de un microbioma “sano” generalmente se centraban en conjuntos de taxones que cabría esperar que se encontraran predominantemente en personas sanas. Si bien los núcleos puramente taxonómicos de cualquier tipo han permanecido esquivos, incluso en poblaciones relativamente estrechamente definidas, cada hábitat de sitio corporal posee fuertes enriquecimientos filogenéticos. Aquí se muestran géneros típicos (o familias en el intestino) en poblaciones sanas en diferentes sitios [7, 9, 15, 33–35]. b Las mediciones metagenómicas han permitido evaluar el potencial funcional del microbioma en diferentes sitios. Estos estudios han producido núcleos funcionales compartidos de manera más consistente de vías específicas de todo el cuerpo y de nicho que se mantienen en la salud [6, 7, 9, 98]. Lipopolisacárido LPS, patrón molecular asociado al patógeno PAMP. c Los patrones de ensamblaje ecológico proporcionan otra posible definición de un microbioma sano, ya que cada huésped puede recurrir a una metapoblación “típica” de microbios potenciales a través de una combinación de procesos parcialmente estocásticos. Estos procesos pueden incluir el orden en que los microbios colonizan su respectivo hábitat humano (afectado por la geografía y las exposiciones tempranas, por ejemplo), la disponibilidad prolongada de cada microbio en el entorno local del huésped y la selección del huésped (a través de la dieta o la genética, adaptada de la Fig. 1 de [101]). d El microbioma saludable también se puede caracterizar en términos de su dinámica, representada aquí en un modelo simplificado como un paisaje energético conceptual. El microbioma infantil (punto amarillo) comienza en un estado inestable y desciende gradualmente hacia uno de los varios estados atractores adultos potencialmente sanos. Las perturbaciones (flechas rojas discontinuas) pueden resistirse (punto verde) o pueden sacar al microbioma del estado saludable, después de lo cual un microbioma resistente volverá a un estado saludable (no necesariamente el estado saludable original) o caerá en un estado no saludable (rojo)
Aunque menos estudiado que el intestino, muchos otros hábitats corporales dentro de individuos sanos están ocupados por comunidades microbianas [7]. La composición de la comunidad es más similar dentro de los hábitats que entre ellos (por ejemplo, las comunidades orales comparten una mayor similitud con las comunidades orales de otras personas que con otros hábitats dentro de la misma persona), aunque, a su vez, las diferencias interindividuales dentro de los hábitats son mucho mayores que la variabilidad intraindividual a lo largo del tiempo [7]. Los sitios orales albergan microbiomas particularmente diversos [33], similares en complejidad al microbioma del intestino [7], y tienden a estar dominados por Streptococcus spp. [7]. Los sitios de la piel difieren principalmente con las propiedades locales de la piel (seca versus húmeda versus sebácea [34]) y están colonizados principalmente por Corynebacterium, Propionibacterium y Staphylococcus [34]. La vagina sana contiene uno de los ecosistemas microbianos más notablemente estructurados, con al menos cinco tipos de comunidad reproducibles, o “tipos de estado de comunidad”, cada uno dominado por una sola especie de Lactobacillus (L. crispatus, L. iners, L. jensenii o L. gasseri) o por una mezcla de otros microbios incluyendo Gardnerella [35, 36]. Los determinantes significativos del tipo de estado comunitario de una mujer incluyen la raza/etnia [35, 37] y el embarazo [37], aunque incluso en este ecosistema estructurado dentro del sujeto la variación longitudinal es sustancial y, hasta la fecha, no tiene causas completamente explicadas.
Varios hábitats corporales importantes tienden a tener una biomasa microbiana particularmente baja en individuos sanos y, por lo tanto, son más difíciles de caracterizar. El pulmón, por ejemplo, es casi estéril en ausencia de infección o enfermedad crónica, lo que genera un gran interés en la identificación de sus residentes normales, pero también desafíos técnicos sustanciales en el muestreo y la secuenciación del sitio [38-40]. Del mismo modo, la leche materna [41] y la placenta [42] son de interés para el establecimiento temprano tanto de un microbioma saludable como de los microbiomas potenciales circulantes de sangre [43] o tejido [44, 45] para el control inmunológico normal de los oportunistas. Existen dificultades considerables en la adquisición de metagenomas de estos entornos y, por lo tanto, la mayoría de los estudios se han basado en estudios de amplicones sensibles a la contaminación [46] y técnicas unicelulares de rendimiento relativamente bajo, como FISH o microfluídica. Por lo tanto, se necesitan estudios a mayor escala cuidadosamente controlados para establecer la funcionalidad de estos desafiantes hábitats microbianos de baja densidad.
Arqueas, virus, hongos y otros eucariotas
El estudio del microbioma saludable se ha enriquecido enormemente para las bacterias [7, 9], con menos atención a otros dominios microbianos. Sin embargo, el microbioma humano abarca el árbol de la vida y, por lo tanto, incluye arqueas, virus y eucariotas. Se ha identificado un pequeño número de géneros de arqueas en el microbioma humano sano, principalmente en el intestino. Las especies del género Methanobrevibacter son las más prevalentes [47] en el intestino, y su estatus como miembros “sanos” de las comunidades de otros sitios del cuerpo sigue siendo algo confuso [48]. Se ha descubierto que Methanobrevibacter smithii, en particular, está bien adaptado al intestino humano, optimizando la digestión de los polisacáridos de la dieta por otros microbios [49] y adaptando su expresión génica en presencia de bacterias intestinales comunes como Bacteroides thetaiotaomicron [49]. El viroma humano es particularmente extenso y, aunque poco caracterizado, se reconoce como una parte integral del ecosistema humano saludable [50]. Con la naturaleza hipervariable de los virus, se espera que cada persona albergue un viroma único [51, 52], que consiste principalmente en bacteriófagos [50] (se estima que el 5 % del complemento génico bacteriano intestinal codifica para las proteínas de los profagos [9]). Los fagos también proporcionan un medio adicional de transferencia horizontal de genes entre bacterias que de otro modo estarían lejanamente relacionadas [53]. Dado que las técnicas de perfiles moleculares para arqueas, virus y eucariotas están aún menos desarrolladas que las de bacterias (incluso las que utilizan enfoques independientes del cultivo [47, 54]), la información sobre la funcionalidad molecular de estos organismos dentro de las comunidades in situ sigue siendo limitada.
Aunque los microorganismos eucariotas más conocidos que se encuentran en el cuerpo humano (principalmente hongos y protistas) suelen ser patógenos, es importante recordar que muchos de estos eucariotas, en particular Candida, Malassezia y Saccharomyces, son omnipresentes incluso en poblaciones sanas [55-58]. Las interacciones entre reinos son responsables de al menos parte del equilibrio ecológico e inmunológico del microbioma sano; por ejemplo, existe una aparente competencia entre bacterias y hongos en los entornos bioquímicos de la piel [59] o en el control de hongos en el intestino [55] y la vagina [60]. Aunque existen pocos ejemplos, se han encontrado relaciones mutualistas directas entre humanos y hongos, de las cuales la mejor caracterizada es la levadura probiótica Saccharomyces boulardii, originalmente aislada para combatir el cólera [61]. Algunos protozoos son incluso habitantes comunes de microbiomas sanos [58, 62], aunque (como los virus) con una variabilidad interpersonal aún mayor que las bacterias [58]. Además, la presencia de algunos protozoos, como el Blastocystis común, se ha asociado con un menor riesgo de enfermedad gastrointestinal [63]. Por último, aunque los eucariotas multicelulares, como los helmintos, se han eliminado generalmente de los microbiomas intestinales en las culturas occidentales, han sido un componente del microbioma intestinal durante una parte significativa de nuestra historia evolutiva reciente [64]. Dadas sus potentes capacidades inmunomoduladoras y sus interacciones con los otros habitantes del microbioma intestinal normal (como los lactobacilos [65]), su eliminación puede haber eliminado un importante educador de nuestro sistema inmunológico [64].
Variación geográfica en el microbioma saludable
Los estudios que contrastan los microbiomas intestinales de diferentes países han identificado diferencias sistemáticas en la composición microbiana, aunque sigue siendo difícil separar los efectos técnicos entre lotes de la biología interpoblacional. La comparación entre las cohortes más grandes de tres continentes (MetaHIT (europea), HMP (estadounidense) y cohortes chinas de diabetes, encontró que la variación entre países en la composición taxonómica superó significativamente la variación interpersonal, lo que no se atribuyó únicamente a las diferencias técnicas en las metodologías experimentales [29]. Sin embargo, estudios internacionales más pequeños también han identificado la geografía como una de las principales fuentes de variación a gran escala en el microbioma, incluso entre América del Norte y del Sur [66], Europa y África [67], Corea y Japón [68], y entre las poblaciones rurales y urbanas de Rusia [69] y China [70]. Entre los posibles impulsores de esta variación, se ha sugerido que la dieta es un factor importante [67], junto con otros factores como la geografía, las exposiciones tempranas y la genética [29, 71]. Ningún estudio ha demostrado aún que ninguno de estos factores sea causal en las grandes diferencias interpoblacionales observadas en los microbiomas sanos [72].
Las diferencias geográficas a nivel de cepa también son de interés, particularmente porque las firmas de cepas exhiben una mayor estabilidad temporal que los perfiles de abundancia microbiana [8, 73, 74]. La investigación en esta área es preliminar, pero muestra que las diferencias de cepas no son particularmente pronunciadas entre países o continentes. Especies como Bacteroides coprocola y Prevotella copri muestran las mayores diferencias [73] y variantes a nivel de cepa en los genes de resistencia a antibióticos que abarcan poblaciones [75]. Los análisis forenses microbianos a nivel de cepa en especies altamente heredables como Helicobacter pylori han sido notablemente perspicaces para rastrear los efectos históricos en el microbioma [76, 77] y las técnicas independientes del cultivo deben aprovecharse para realizar estudios poblacionales exhaustivos a gran escala en el futuro.
Establecimiento del microbioma y colonización temprana
Los factores que influyen en la dinámica del microbioma en los primeros años de vida son importantes precipitadores de un microbioma sano. La introducción y persistencia microbiana es un proceso semiestocástico influenciado por muchos elementos (Fig. 1c), produciendo una configuración saludable similar a la de un adulto solo después de los primeros años de vida [66, 78-80]. El enriquecimiento del microbioma intestinal infantil para simbiontes como Bacteroides, Parabacteroides, Clostridium, Lactobacillus, Bifidobacterium y Faecalibacterium prausnitzii proporciona varios determinantes de un microbioma saludable. Una vez establecidos, estos son los principales productores de ácidos grasos de cadena corta (AGCC), una importante fuente de energía a partir de carbohidratos no digeribles [81]. Los AGCC son inmunomoduladores [82], inhiben patógenos comunes y se supone que poseen propiedades supresoras de tumores [83, 84]. El microbioma intestinal es un requisito inextricable para la educación del sistema inmunitario y el establecimiento de estos géneros beneficiosos en una etapa temprana de la vida promueve la tolerancia inmunitaria y, en consecuencia, puede atenuar o derogar las enfermedades autoinmunes [1, 85-89].
El modo de parto puede afectar al establecimiento de la microbiota en los primeros años de vida, de modo que la cesárea se asocia con el enriquecimiento de los oportunistas, como Haemophilus spp., Enterobacter cancerogenus/E. hormaechei, Veillonella dispar/V. parvula [78] y Staphylococcus [80]. Estos microbios continúan persistiendo al menos durante el primer año de vida [78] y posiblemente contribuyen a la carga de infección infantil. La dieta también representa una fuerte presión selectiva sobre el microbioma [71, 90] y la lactancia materna (como primera dieta) favorece ciertos clados microbianos de entre la microbiota inicial que pueden haberse ensamblado al azar. Por ejemplo, los oligosacáridos de la leche humana (HMO) pueden ser utilizados como única fuente de carbono por solo un puñado de especies de Bifidobacterium y Bacteroides [91] y, más aún, recientemente se demostró que los oligosacáridos de la leche bovina (BMO) promueven el crecimiento y el metabolismo de una manera dependiente de la microbiota en modelos animales infantiles [92]. Si bien es posible que este modelo no se traduzca directamente a los bebés humanos debido a la diversidad estructural única, la complejidad y la alta concentración de HMO [93, 94], respalda aún más la inferencia de que los beneficios a largo plazo de la lactancia materna [95] están mediados, en parte, por el microbioma.
Señas de identidad de la salud
Núcleo funcional
Si bien se observan grandes diferencias interpersonales en la composición taxonómica del microbioma en todos los sitios, la abundancia de vías metabólicas es considerablemente más consistente entre las personas para un sitio determinado [7, 9, 26, 27]. Además, si bien la composición del microbioma cambia drásticamente durante los primeros años de vida, este perfil funcional se establece desde el principio y permanece estable a partir de entonces, al menos en el intestino [72]. Esto sugiere que una definición de un microbioma saludable “central” podría incluir combinaciones específicas de familias de genes microbianos, módulos metabólicos y vías reguladoras que, en conjunto, promueven una ecología estable asociada al huésped [96, 97]. Este núcleo incluye funciones de al menos tres grupos: en primer lugar, y más simplemente, las funciones de mantenimiento necesarias para toda la vida microbiana, como la transcripción y la traducción, la producción de energía y los componentes estructurales [6, 7, 9]. En segundo lugar, este núcleo incluye procesos que son específicos de los microbiomas asociados al ser humano en los hábitats corporales, como la adhesión a las superficies celulares del huésped y la producción de compuestos implicados en la interacción huésped-microbio (incluidas las vitaminas esenciales, como la vitamina K, y los compuestos inmunoestimulantes) [6, 7]. Por último, cada uno de los diferentes hábitats corporales tiene sus propias funciones básicas especializadas [98]. Por ejemplo, en el intestino, las funciones básicas incluyen la biodegradación de glicosaminoglicanos, la producción de varios ácidos grasos de cadena corta, el enriquecimiento de lipopolisacáridos específicos y la producción de vitaminas y aminoácidos esenciales [6, 9, 98, 99] (Fig. 1b). Cuáles de estas funciones tienden a enriquecerse en una población determinada puede verse afectada por presiones selectivas a largo plazo, como la dieta [67]. Por lo tanto, una condición necesaria para un microbioma saludable es la presencia de un conjunto de especies microbianas que puedan llevar a cabo conjuntos específicos de funciones biomoleculares en cada uno de los entornos bioquímicos específicos del nicho en todo el cuerpo.
Ecología comunitaria saludable
Si las comunidades microbianas se reúnen sobre la base de su cobertura de un conjunto básico de funciones mientras seleccionan entre una gran metapoblación de colonizadores potenciales, es probable que sean ecológicamente diversas [100-102], tanto en términos de riqueza (número de taxones presentes) como de uniformidad (abundancia de muchos constituyentes microbianos). La alta diversidad se ha asociado generalmente con la salud [11] y la estabilidad temporal [103]. Esto último podría, por ejemplo, ser el resultado de la mayor redundancia funcional que viene con un conjunto más diverso de microbios, incluso si el potencial funcional del ensamblaje es mínimamente alcanzable con menos taxones. Por el contrario, se observa una relativa falta de diversidad en el microbioma intestinal en enfermedades que van desde la obesidad [26] hasta la enfermedad inflamatoria intestinal [104] y la diabetes tipo 1 [72] y 2 [28]; y en el microbioma cutáneo en dermatitis atópica [105] y psoriasis [106]. Los antibióticos también causan una reducción drástica en la diversidad del microbioma con una dinámica de recuperación muy variable [107], lo que podría debilitar la capacidad de la comunidad para excluir patógenos. Esto puede despejar el camino para la infección por patobiontes, miembros normales de la comunidad microbiana que se vuelven perjudiciales bajo la perturbación, como Candida albicans [57]. Sin embargo, el principio de que una alta diversidad es “saludable” no se aplica a todos los sitios del cuerpo, ya que la diversidad en el microbioma vaginal puede asociarse con vaginosis bacteriana [108], neoplasia intraepitelial cervical [109] (un crecimiento anormal en el cuello uterino), parto prematuro [36] e inflamación [110].
Dada la observación típica del aumento de la diversidad del microbioma en la salud, se ha planteado la hipótesis [111] de que la diversidad microbiana intestinal consistentemente reducida de los países desarrollados puede explicar tasas de enfermedades crónicas más altas en relación con las observadas en los países en desarrollo y las sociedades primitivas [66, 112, 113], denominada la “hipótesis del microbioma que desaparece” [111]. Esta pérdida de diversidad puede estar relacionada con una dieta alta en grasas, alta en azúcar refinada y baja en fibra [114]. Los ratones humanizados con una dieta de este tipo exhiben un agotamiento en la diversidad microbiana [114] y, aunque esto es recuperable al volver a una dieta alta en fibra dentro de una generación, se fija después de cuatro generaciones [114]. Si este resultado se generaliza a las poblaciones humanas, aumenta la urgencia de desarrollar métodos terapéuticos o de mantenimiento del microbioma dirigidos racionalmente, a fin de dirigir los microbiomas menos beneficiosos para la salud hacia ensamblajes más naturales. La hipótesis del microbioma que desaparece representa de alguna manera una evolución de las hipótesis de la “higiene” o de los “viejos amigos” [115], todas las cuales sugieren que, si bien las cohortes modernas de América del Norte o Europa pueden representar microbiomas “saludables”, su relación con lo que es evolutivamente “normal” puede ser más compleja.
Resistencia, resiliencia y estabilidad
Otras características distintivas de la salud desde la perspectiva de la ecología microbiana son la capacidad de resistir la perturbación (que puede resultar de la entrada de un patógeno, la alteración de la dieta o la medicación) y volver a un estado saludable después. Estas propiedades se han denominado resistencia y resiliencia, respectivamente [2]. Por ejemplo, después de un tratamiento con antibióticos, las comunidades intestinales sanas generalmente se recuperan a su estado anterior después de unas pocas semanas o meses [116]. Por lo tanto, una definición reciente de salud microbiana no comprende explícitamente un único estado estático, sino más bien un equilibrio dinámico [2]. Desde este punto de vista, un microbioma sano corresponde a un atractor de un sistema dinámico subyacente (Fig. 1d), de manera similar al destino celular en un metazoo [117]. Los atractores capturan tanto la resistencia como la resiliencia, en el sentido de que el sistema resistirá una desviación de un atractor, y a menos que una fluctuación (que podría deberse a una perturbación externa o a la estocasticidad interna) sea lo suficientemente grande, tenderá a volver al área de estado estacionario [117]. Los ejemplos más visibles en el microbioma humano pueden ser las transiciones entre los tipos de estado comunitario en la vagina sana; Aunque todavía no se han enumerado sus implicaciones sanitarias específicas, no todos los tipos de Estado comunitario tienen el mismo grado de estabilidad [36]. El microbioma intestinal también está cambiando, ganando y perdiendo especies con el tiempo, con diferentes taxones que tienen diferentes estabilidades y algunos permanecen constantemente en el intestino durante muchos años [8]. Los mecanismos por los cuales persisten taxones específicos aún no están bien delineados, pero es interesante especular si tales mecanismos podrían estar relacionados con los principios impulsores detrás del ensamblaje del microbioma. Si las comunidades específicas se reúnen principalmente para llenar un conjunto de nichos funcionales adecuados para el hábitat [6], entonces las especies que proporcionan funciones metabólicas, de señalización, inmunomoduladoras u otras funciones clave en un conjunto particular pueden ser más estables temporalmente que las de la periferia funcional. El acoplamiento de la dinámica con la diversidad taxonómica y el inmenso potencial funcional molecular del microbioma es, por tanto, un recordatorio de la complejidad del microbioma humano y, en consecuencia, de la dificultad de definir incluso el concepto aparentemente simple de salud microbiana.
Perspectiva
La era de la epidemiología del microbioma completo a escala poblacional ha comenzado recientemente, con el HMP [7, 118] y el MetaHIT [9, 29] entre las primeras cohortes grandes en incluir datos de referencia amplios en salud, y varias cohortes más que pronto llegarán. Hasta la fecha, los datos han estado dominados por estudios transversales basados en amplicones de poblaciones occidentales, todos los cuales son eficientes y accesibles, pero que aún no pintan una imagen coherente y completa del microbioma global, dinámico y saludable. La epidemiología a gran escala en otras áreas de la salud humana, como la nutrición y el estilo de vida, ha construido una base sólida para cohortes prospectivas y de larga duración, análisis minuciosos e instrumentos de medición cuidadosamente validados [119-121], todos los cuales representan vías de exploración particularmente prometedoras para el microbioma. La anidación de estudios longitudinales de microbioma en cohortes existentes tiene la ventaja de utilizar información sobre el estilo de vida, la dieta, la medicina y el fenotipo recopilada a largo plazo, así como la integración con muestras biológicas almacenadas. Un ejemplo de un estudio no convencional a gran escala, notable por su infraestructura y alcance, es el proyecto American Gut: una fuente financiada por crowdfunding de datos de referencia del microbioma junto con metadatos ambientales proporcionados por el sujeto. Los estudios prospectivos con datos moleculares detallados, aunque más costosos y logísticamente desafiantes, también serán necesarios para facilitar los modelos predictivos y establecer la causalidad de las disbiosis. El proyecto en curso “HMP2” o Proyecto Integrativo del Microbioma Humano (iHMP, por sus siglas en inglés) [122] incluye tres estudios longitudinales de este tipo, que están proporcionando datos multiómicos para la salud y las enfermedades crónicas, junto con protocolos y herramientas computacionales como base para trabajos futuros.
Si bien muchos estudios actuales sobre el microbioma se centran en la enfermedad, una mejor comprensión del microbioma saludable ayudará a desarrollar nuevos diagnósticos y terapias de la comunidad microbiana [123]. En la medida en que se puedan definir las características universales del microbioma sano, su ausencia puede ser predictiva de la aparición de la enfermedad en general, al igual que la presencia de características específicas de la disbiosis de cualquier afección (especialmente útil si ocurre antes de la aparición de la enfermedad). Alternativamente, la medicina personalizada y el monitoreo longitudinal pueden servir para el mismo propósito con respecto a la salida del propio estado “saludable” de un individuo [1, 104]. Desde el punto de vista terapéutico, a medida que se desarrollan intervenciones dirigidas a manipular el microbioma, el tratamiento de una disbiosis no tiene por qué volver al estado saludable del que partió un individuo (debido a una perturbación como el tratamiento con antibióticos o la invasión de un patógeno), sino quizás sólo a un estado sano (Fig. 1d). Del mismo modo, incluso si una disbiosis microbiana demuestra ser sensible en lugar de causal en cualquier estado de enfermedad dado, el retorno a un estado “saludable” aún puede proporcionar un beneficio terapéutico [73, 101, 124].
Una de las mayores lagunas pendientes en la comprensión de la biología básica del microbioma “sano” se encuentra quizás en el nivel de la anotación de su función molecular: hasta el 50 % de las familias de genes microbianos que se encuentran en el microbioma humano permanecen funcionalmente sin caracterizar, incluso en entornos bien estudiados como el intestino [9, 25, 29]. Esto también es cierto hasta cierto punto en los genomas de aislados microbianos individuales, donde incluso la bien estudiada E. coli K12 contiene alrededor del 18 % de productos génicos sin función reportada [125], con una cantidad apreciablemente mayor a nivel del pangenoma de la especie de E. coli [126]. Es probable, por ejemplo, que algunos de estos genes sean responsables de las interacciones microbio-microbio o huésped-microbio y, por lo tanto, solo se expresen o caractericen en entornos comunitarios. Los estudios a escala poblacional del microbioma pueden utilizarse para mitigar parcialmente esta situación, en el sentido de que se puede priorizar la caracterización de las familias de genes microbianos que son prevalentes y abundantes, pero que aún no se comprenden bien. Del mismo modo, las comunidades microbianas proporcionan una nueva fuente de información de culpabilidad por asociación que se puede utilizar computacionalmente para generar predicciones de la función de los genes [127, 128]. Sin embargo, volver a las raíces microbiológicas del campo puede resultar en última instancia más importante en esta área: las mejores caracterizaciones bioquímicas aún se derivan de la fisiología basada en cultivos, el metabolismo microbiano, el cocultivo y las interacciones, y los entornos de laboratorio controlados junto con ensayos moleculares de alto rendimiento [15, 129, 130].
Los estudios sobre el microbioma, tanto en la salud como en la enfermedad, deben continuar integrando la epidemiología a escala poblacional con estudios clínicos estrechos pero profundos en el entorno de la medicina personalizada. En ambos casos, los estudios del microbioma de todo el cuerpo pueden verse como una extensión de las técnicas microbianas ya utilizadas para la vigilancia de enfermedades infecciosas [131]: en lugar de esperar a monitorear el brote de un patógeno en una población o su persistencia dentro de un individuo, nuestra comunidad microbiana completa podría ser monitoreada para el mantenimiento de la salud o las salidas a la enfermedad. Esto es igualmente cierto en la integración de la actividad del microbioma con el estado inmunológico, transcripcional, epigenético y clínico del huésped: la medicina comunitaria microbiana de precisión debe basarse en las interacciones huésped-microbioma como un componente clave. Esto ayudará a identificar rápidamente los patógenos potenciales [132] y permitirá determinar las intervenciones “correctas” para restaurar la salud después de la disbiosis, que van desde cambios en la dieta o el estilo de vida, pasando por probióticos, hasta productos farmacéuticos dirigidos microbianamente [133]. Por lo tanto, una mejor comprensión del microbioma saludable debe abordarlo como un aspecto de la salud personalizada profundamente monitoreada (por ejemplo, [121]) y debe integrar la evaluación a escala poblacional de la comunidad microbiana con una comprensión molecular bien caracterizada y análisis de cómo se mantienen los estados beneficiosos de la comunidad en todo el cuerpo y durante toda la vida.
Abreviaturas
PESCADO:
hibridación fluorescente in situHMO:
oligosacáridos de la leche humanaHMP:
Proyecto Microbioma HumanoMetaHIT:
Metagenomas del Tracto Intestinal HumanoAGCC:
Ácido graso de cadena corta

